Présentation, installation¶
Qu’est-ce que Numba ?¶
Numba est un module Python utilisé pour du calcul intensif et qui permet, sous réserve de travailler avec des tableaux Numpy :
- d’améliorer considérablement les performances de certains programmes en gardant une syntaxe presque identique au programme n’utilisant pas Numba
- de donner accès, avec une syntaxe légère, à des boucles parallèles sur un processeur multicœur,
- de donner accès, sous Python, à l’environnement Cuda de programmation sur GPU, avec bien sûr, d’importantes améliorations de performances par rapport au calcul sur un simple CPU, même multicœur.
Il y a toutefois de nombreuses contreparties à ces excellentes performances.
Guide de lecture¶
Ce document sur le module Numba est divisé est 5 parties mais si vous voulez aller à l’essentiel, voici ce que je vous recommande :
Commencez par lire : Utilisation de Numba sur Google Colab
Ensuite, il faut avoir parcouru le paragraphe : Les deux modes de fonctionnement de Numba
Vous pouvez alors directement lire la partie Programmation Numba sur CPU. L’essentiel se trouve dans les paragraphes suivants :
Lire le paragraphe Utiliser Numba avec un GPU sous Google Colab et, éventuellement, les deux paragraphes de généralités sur le GPGPU avec Numba et les cartes graphiques.
Lire la dernière partie Programmation Numba sur GPU.
La partie sur les structures de données Numba a un intérêt limité dans la mesure où elles sont expérimentales et d’une efficacité variable.
Les autres parties sont consacrées à l’installation et à des informations diverses sur Numba.
Ce document n’a pas un caractère approfondi et propose essentiellement un panorama des fonctionnalités de Numba.
Je n’explique pas comment utiliser des feuilles Jupyter Notebook. Sous Linux ou macOS, je suppose que vous avez l’habitude de travailler en ligne de commande. Je suppose aussi que vous avez utilisé pip (appelé pip3 sur mon système Linux).
Pouvoir utiliser Numba¶
Accès à Numba¶
Pour pouvoir utiliser pleinement Numba (sous-entendu : avec GPU si disponible), vous avez les possibilités suivantes :
utiliser Google Colab. Avantages :
- fontionne d’emblée, rien à installer
- on peut utiliser des GPU presque immédiatement
utiliser Numba via le toolkit Anaconda 3 Personal Edition (Linux, macOS, fortement recommandé pour Windows)
installer Numba via pip (linux, macOS)
utiliser Numba en ligne avec mybinder, cf. la section dédiée ci-dessous.
Version¶
Pour connaître la version de Numba que vous utilisez, il suffit d’écrire le code suivant :
import numba
print(numba.__version__)
qui affiche chez moi (mai 2021) :
0.53.1
Le problème des GPU¶
L’installation par défaut d’Anaconda ou l’installation via pip ne suffisent pas à exploiter votre (éventuel) GPU sous Numba. Si vous tentez de l’utiliser, vous aurez différents types de message d’erreur et qui dépendent de l’état de votre système.
Par exemple, sous Anaconda ou avec pip, le simple fichier suivant
from numba import cuda
cuda.detect()
pourrait renvoyer l’erreur suivante :
CudaSupportError: Error at driver init:
CUDA driver library cannot be found.
If you are sure that a CUDA driver is installed,
try setting environment variable NUMBA_CUDA_DRIVER
with the file path of the CUDA driver shared library
(ici, le driver matériel n’a pas été installé).
Utilisation de Numba sur Google Colab¶
Google Colab est un service gratuit mis à la disposition d’un utilisateur ayant un compte Google et permettant de programmer dans des feuilles Jupyter Notebook et exécutées sur l’infrastructure de Google.
Je suppose que
- vous avez installé l’application Google Colaboratory sur votre Google-Drive
- vous avez déjà codé en Python dans des feuilles Jupyter Notebook.
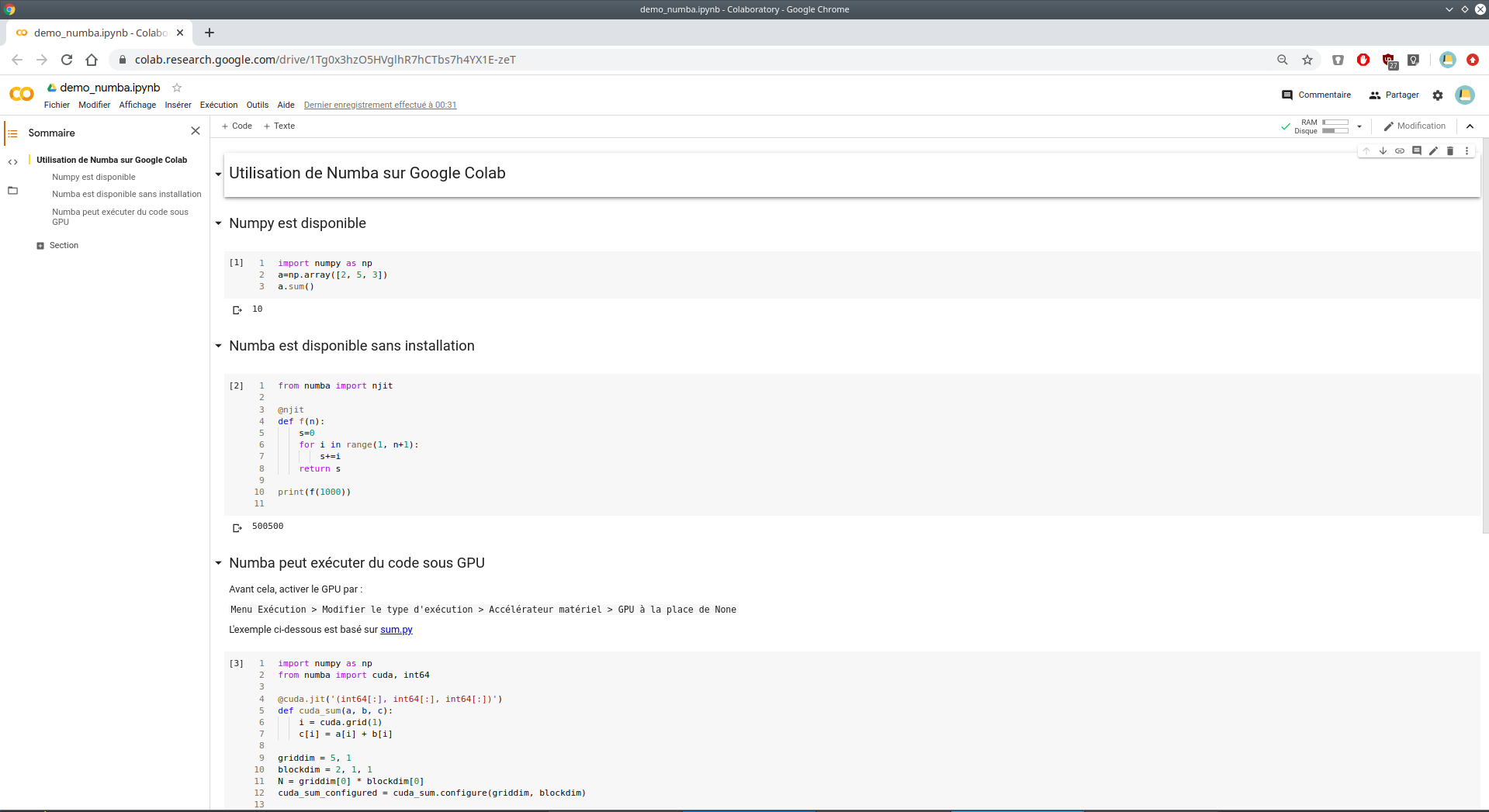
La feuille que nous allons écrire ressemblera à ça :
et est disponible et exécutable en ligne ICI.
Modules disponibles¶
Les feuilles Google Colab sont destinées à faire des data sciences. En particulier, par défaut, les modules Python indispensables, comme Numpy ou SciPy, sont disponibles sans besoin de les installer. Par exemple, vous pouvez directement écrire dans une cellule du code comme
import numpy as np
a=np.array([2, 5, 3])
a.sum()
qui affichera
10
Mais, mieux que cela, Numba est disponible par défaut, autrement dit, déjà installé. Donc vous pouvez directement écrire un code tel que
from numba import njit
@njit
def f(n):
s=0
for i in range(1, n+1):
s+=i
return s
print(f(1000))
et qui affichera
500500
Version disponible¶
Testons la version utilisée :
import numba
numba.__version__
0.51.2
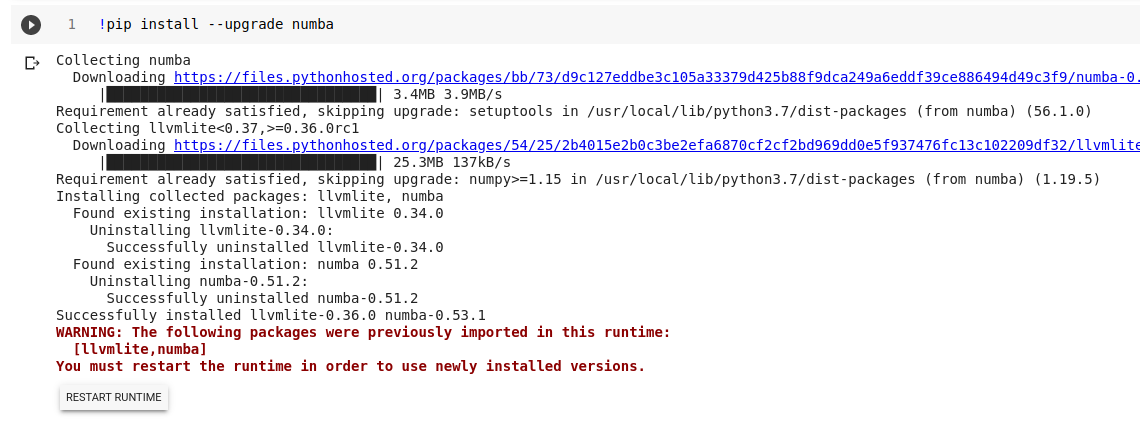
Pour le coup, ce n’est pas la dernière disponible qui est, en mai 2021, la version 0.53. On essaye de mettre à jour avec une commande spéciale
!pip install --upgrade numba
(sans omettre le point d’exclamation) qui affiche
Après redémarrage de l’environnement d’exécution effectué en cliquant sur le bouton qui est apparu (Restart Runtime), si on teste :
import numba
numba.__version__
on voit que Numba a été mis à jour :
0.53.1
Pour l’usage des GPU sous Google Colab, voir le paragraphe dédié dans la partie correspondante.
En outre, il est possible, avec des commandes spécifiques d’installer la plupart des bibliothèques d’apprentissage non déjà installées (voir un exemple avec Cupy dans la dernière partie).
Installation de Numba avec pip¶
Ce qui suit s’applique à Windows, Linux et masOS. Utiliser pip pour installer Numba est la méthode la plus simple. Bien que cette installation ne suffise pas à utiliser un éventuel GPU, elle délivre déjà une bonne partie de la puissance de Numba.
Il suffit d’utiliser pip en ligne de commande de la manière suivante (ici sous Linux) :
po@po-18:~$ pip3 install numba
Collecting numba
Downloading https://files.pythonhosted.org/packages/73/c1/
7e8bf88ee00aa89270c364c22c3f21a1e23243cbcaf3f0714fd5af953
a34/numba-0.50.1-cp36-cp36m-manylinux1_x86_64.whl (2.6MB)
100% |████████████████| 2.6MB 731kB/s
Collecting llvmlite<0.34,>=0.33.0.dev0 (from numba)
Downloading https://files.pythonhosted.org/packages/d5/37/
10d2c4ba0c131cf112d0a8fc97af7a77ca4a769aff626b2bfa2e57e679
cf/llvmlite-0.33.0-cp36-cp36m-manylinux1_x86_64.whl (18.3MB)
100% |████████████████| 18.3MB 104kB/s
Collecting setuptools (from numba)
Downloading https://files.pythonhosted.org/packages/8e/11/
9e10f1cad4518cb307b484c255cae61e97f05b82f6d536932b1714e01b
47/setuptools-49.2.0-py3-none-any.whl (789kB)
100% |████████████████| 798kB 2.4MB/s
Collecting numpy>=1.15 (from numba)
Downloading https://files.pythonhosted.org/packages/22/e7
/4b2bdddb99f5f631d8c1de259897c2b7d65dcfcc1e0a6fd17a7f62923
500/numpy-1.19.1-cp36-cp36m-manylinux1_x86_64.whl (13.4MB)
100% |████████████████| 13.4MB 143kB/s
Installing collected packages: llvmlite, setuptools, numpy, numba
Successfully installed llvmlite-0.33.0 numba-0.50.1 numpy-1.19.1
setuptools-49.2.0
Vous noterez que l’installation de Numba entraîne l’installation de Numpy.
Une fois Numba installé, testez en exécutant sous Python le programme suivant
tester_numba.py
from numba import njit
@njit
def f(n):
s=0
for i in range(1, n+1):
s+=i
return s
print(f(1000))
et qui doit afficher
500500
On peut vérifier la version :
Python 3.6.9 (default, Jul 17 2020, 12:50:27)
[GCC 8.4.0] on linux
Type "help", "copyright", "credits" or "license" for more information.
>>> import numba
>>> numba.__version__
'0.50.1'
>>>
Mise-à-jour¶
Pour mettre à jour une version de Numba, utiliser en ligne de commande (testé sous Linux seulement) :
$ pip3 install --upgrade numba
WARNING: pip is being invoked by an old script wrapper. This will fail in a future version of pip.
Please see https://github.com/pypa/pip/issues/5599 for advice on fixing the underlying issue.
To avoid this problem you can invoke Python with '-m pip' instead of running pip directly.
Defaulting to user installation because normal site-packages is not writeable
Requirement already satisfied: numba in /home/po/.local/lib/python3.6/site-packages (0.50.0)
Collecting numba
Downloading numba-0.53.1-cp36-cp36m-manylinux2014_x86_64.whl (3.4 MB)
|████████████████████████████████| 3.4 MB 3.5 MB/s
Requirement already satisfied: numpy>=1.15 in /home/po/.local/lib/python3.6/site-packages (from numba) (1.19.1)
Requirement already satisfied: setuptools in /home/po/.local/lib/python3.6/site-packages (from numba) (56.0.0)
Collecting llvmlite<0.37,>=0.36.0rc1
Downloading llvmlite-0.36.0-cp36-cp36m-manylinux2010_x86_64.whl (25.3 MB)
|████████████████████████████████| 25.3 MB 20.3 MB/s
Installing collected packages: llvmlite, numba
Attempting uninstall: llvmlite
Found existing installation: llvmlite 0.33.0
Uninstalling llvmlite-0.33.0:
Successfully uninstalled llvmlite-0.33.0
Attempting uninstall: numba
Found existing installation: numba 0.50.0
Uninstalling numba-0.50.0:
Successfully uninstalled numba-0.50.0
Successfully installed llvmlite-0.36.0 numba-0.53.1
Utilisation d’Anaconda¶
Anaconda Individual Edition est une suite gratuite et open-source d’outils (je dirai parfois toolkit) permettant de faire des data sciences en Python. Elle facilite en particulier l’installation et la maintenance des outils utilisés.
En installant Anaconda 3, vous pourrez utiliser pleinement Numba, y compris en faisant appel à un GPU si votre machine en dispose.
Se rendre sur la page de téléchargement du toolkit et téléchargez la version en fonction de votre OS (le fichier a une taille autour de 500 Mo). Pour macOS, choisir le version en ligne de commande.
L’installation est expliquée ci-dessous selon l’OS que vous utilisez.
Dès qu’Anaconda est installé, Numba est accessible dans sa version CPU. Pour utiliser le GPU (si disponible sur votre machine), il faudra compléter l’installation (4e partie du document).
Installation d’Anaconda sous Linux ou macOS¶
La version d’Anaconda que vous aurez téléchargée est un gros script shell ayant un nom tel que :
Anaconda3-2020.07-Linux-x86_64.sh
(pour macOS, il faut avoir téléchargé l’installeur en ligne de commande).
Ce qui suit a été réalisé sous Ubuntu versions 18 et 20, c’est analogue sous macOS. L’installation dure autour de 5 minutes en principe. En ligne de commande, se placer dans le répertoire où se trouve le fichier ci-dessus et lancer la commande
$ bash Anaconda3-2020.07-Linux-x86_64.sh
Cela va installer Anaconda 3 dans le répertoire type /home/moi/anaconda3. Il installe énormément d’outils (des dizaines voire des centaines), en particulier toutes les bibliothèques utilisées en apprentissage automatique. Toute l’installation se fait en mode texte mais vous n’aurez qu’à répondre à des questions en tapant yes (si vous êtes d’accord).
A la dernière étape de l’installation, l’installeur va vous demander s’il doit initialiser Anaconda avec la commande conda init. Si vous répondez yes, Anaconda modifiera votre fichier caché bashrc avec les conséquences suivantes :
- Anaconda deviendra votre installation de Python par défaut (au lieu de l’installation système)
- le prompt de votre terminal sera modifié dans toutes ses utilisations (il sera précédé de (base), voir ci-dessous)
- vous aurez accès à tous les outils d’Anaconda directement depuis votre console.
Plus précisément, votre bash aura la forme suivante :
(base) po@po-ta:~$
Personnellement, je réponds no car on peut accéder aux possibilités d’Anaconda en passant par leur navigateur maison.
Au passage, il faut savoir que par défaut, Anaconda installe Python 3.8 et pour le lancer on tape juste python, sans numéro (y compris sur les versions d’Ubuntu antérieures à la 20.04).
Pour faire des essais ou si vous savez ce que vous faites, vous pouvez répondre yes. De toutes façons, vous pouvez toujours revenir en arrière en supprimant du fichier bashrc ce qu’Anaconda a rajouté, à savoir le code (équivalent au) suivant :
# >>> conda initialize >>>
# !! Contents within this block are managed by 'conda init' !!
__conda_setup="$('/home/po/anaconda3/bin/conda' \
'shell.bash' 'hook' 2> /dev/null)"
if [ $? -eq 0 ]; then
eval "$__conda_setup"
else
if [ -f "/home/po/anaconda3/etc/profile.d/conda.sh" ]; then
. "/home/po/anaconda3/etc/profile.d/conda.sh"
else
export PATH="/home/po/anaconda3/bin:$PATH"
fi
fi
unset __conda_setup
# <<< conda initialize <<<
Pour revenir en arrière, il vous suffit de le supprimer du fichier bashrc ou de le commenter en mettant des caractères dièse devant chaque ligne, comme ceci :
# >>> conda initialize >>>
# !! Contents within this block are managed by 'conda init' !!
# __conda_setup="$('/home/po/anaconda3/bin/conda' \
# 'shell.bash' 'hook' 2> /dev/null)"
# if [ $? -eq 0 ]; then
# eval "$__conda_setup"
# else
# if [ -f "/home/po/anaconda3/etc/profile.d/conda.sh" ]; then
# . "/home/po/anaconda3/etc/profile.d/conda.sh"
# else
# export PATH="/home/po/anaconda3/bin:$PATH"
# fi
# fi
# unset __conda_setup
# <<< conda initialize <<<
Plus simple encore, vous pouvez désactiver le shell conda par :
(base) po@po-ta:~$ conda deactivate
po@po-ta:~$
mais cela n’aura un effet que dans le terminal où vous aurez tapé la commande.
Pour lancer le navigateur d’Anaconda, normalement vous tapez
$ anaconda-navigator
Si comme moi, vous n’avez pas demandé à l’installeur de changer votre bashrc, il faut écrire en console le nom complet de l’exécutable, à savoir :
$ ~/anaconda3/bin/anaconda-navigator
Si vous êtes amené à répéter ce genre de commande souvent, autant écrire dans votre bashrc un alias, par exemple :
alias anav="~/anaconda3/bin/anaconda-navigator"
et vous n’aurez plus qu’à taper anav pour lancer la navigateur.
Quand vous lancez le navigateur (temps de chargement assez long), il a l’allure suivante :
Vous pouvez alors utiliser différents éditeurs (Jupyter Notebook, Spyder, VSC) en cliquant sur le bouton Launch pour écrire, exécuter, sauvegarder votre code Python et qui sera exécuté par le toolkit.
Installation d’Anaconda sous Windows 10¶
Pour disposer de Numba, je vais utiliser la suite Anaconda 3 qu’il faut avant tout, installer.
Installation d’Anaconda 3 Individual Edition¶
Se rendre ICI et télécharger la version pour Windows. Installez. C’est assez long (compter 10 bonnes minutes), la barre de progression n’est pas pertinente.
Il installe énormément d’outils (des dizaines voire des centaines), en particulier toutes les bibliothèques utilisées en apprentissage automatique. Vous n’avez quasiment rien à faire et je vous suggère d’accepter les options par défaut, en particulier qu’Anaconda devienne votre installation de Python par défaut, car cela simplifie beaucoup les installations. A la fin de l’installation, Anaconda aura installé dans mon répertoire C:\Users\po\anaconda3 quelques 130 000 fichiers sur 5 Go !
Au passage, il faut savoir que par défaut, Anaconda installe Python 3.8 et pour le lancer en ligne de commande (si cela vous arrive) on tape juste python, sans numéro.
A la fin de l’installation, une entrée est crée dans le menu Démarrer. Pour utiliser Anaconda, le plus simple est de lancer leur navigateur, il s’appelle Anaconda-Navigator.
Quand vous lancez le navigateur (temps de chargement assez long), il a l’allure suivante :
Vous pouvez alors utiliser différents éditeurs (Jupyter Notebook, Spyder, VSC) en cliquant sur le bouton Launch pour écrire, exécuter, sauvegarder votre code Python et qui sera exécuté par le toolkit.
La désinstallation d’Anaconda est très très longue (tant il installe de fichiers).
Utiliser Numba sous Windows avec Anaconda¶
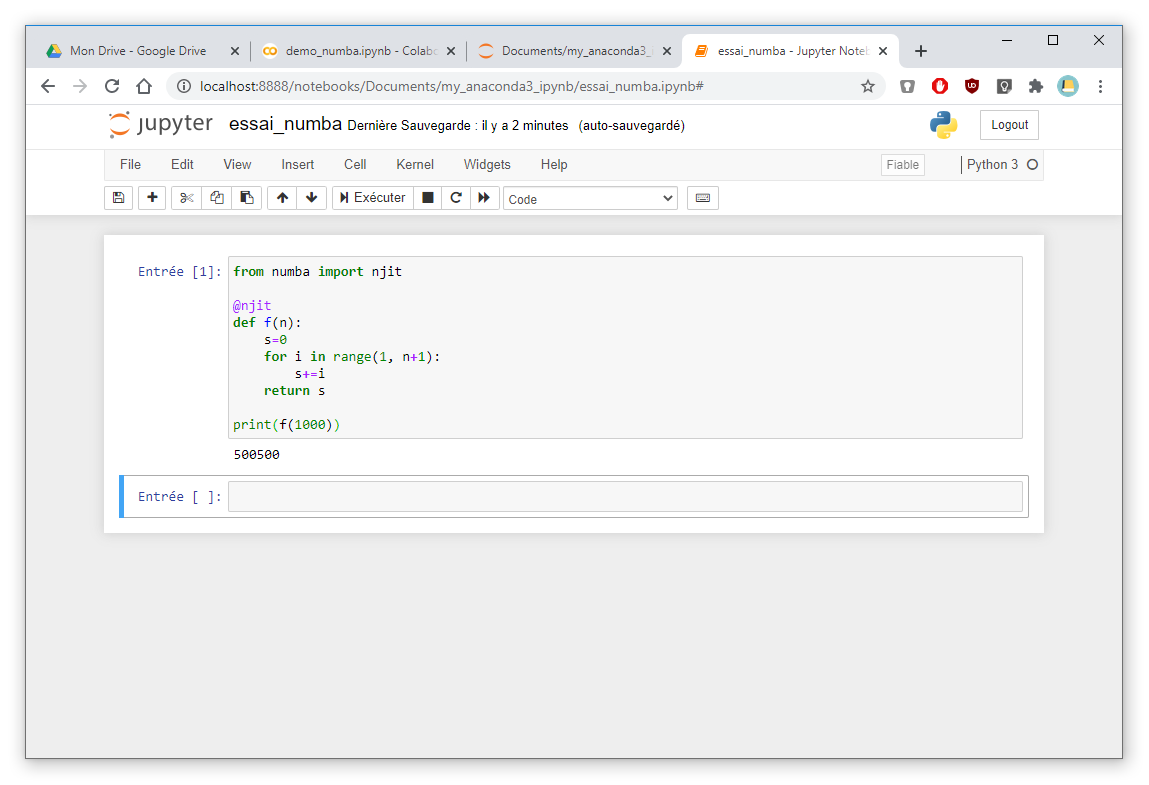
Quand vous installez Anaconda, bonne nouvelle : Numba est déjà installé (mais cette installation permet uniquement un usage sans GPU). Pour tester, il suffit d’ouvrir une feuille Jupyter Notebook (qu’Anaconda a installé aussi) :
Menu Démarrer > Anaconda3 > Jupyter Notebook
Créez une feuille de travail (cliquer sur le bouton New), dans mon cas essai_numba.ipynb, et dans une cellule de code, placez le code :
1 2 3 4 5 6 7 8 9 10 | from numba import njit
@njit
def f(n):
s=0
for i in range(1, n+1):
s+=i
return s
print(f(1000))
|
Il utilise Numba (cf. ligne 1). Exécutez le code en cliquant sur le triangle dans la barre d’outils et vérifiez que la sortie est :
500500
La feuille apparaît sous cette forme :
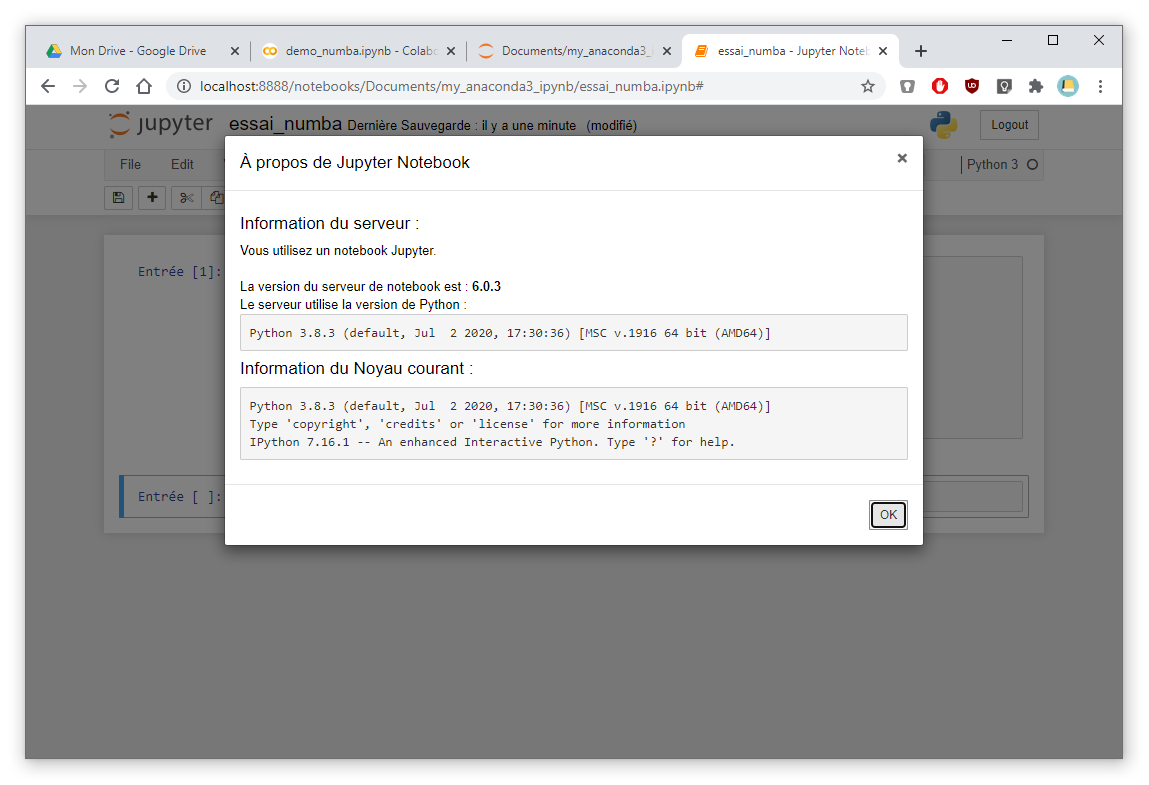
Au passage, on peut vérifier la version de Jupyter Notebook, de python et de IPython :
Utiliser Numba via Anaconda sous Linux¶
Anaconda 3 installe par défaut Numba (mais cette installation permet uniquement un usage sans GPU). Nous allons faire un test avec le fichier suivant :
tester_numba.py
from numba import njit
@njit
def f(n):
s=0
for i in range(1, n+1):
s+=i
return s
print(f(1000))
Pour comprendre ce qui suit, il faut avoir lu la partie sur l’installation d’Anaconda.
Prompt sous Anaconda¶
Si votre bashrc a été modifié par Anaconda au moment de l’installation, vous faites comme vous faisiez pour éxécuter un fichier Python avant l’installation d’Anaconda. Ce qui donne par exemple :
(base) po@po-ta:~$ python tester_numba.py
500500
(base) po@po-ta:~$
Les deux modes de fonctionnement de Numba¶
Le code auquel Numba s’applique s’exécute sous deux modes :
- soit le mode object,
- soit le mode nopython.
C’est uniquement le second mode qui est susceptible d’apporter des gains de performances et donc le premier mode ne sera pas du tout utilisé dans ce document.
En outre, l’efficacité du mode nopython sur des structures de données tient à l’utilisation quasi-obligatoire de tableaux Numpy même s’il faut parfois nuancer quand on utilise des structures de données propres à Numba.
Lorsque le programme s’y prête, Numba produit un code qui s’exécute à la vitesse du C++, parfois même plus rapide. Et le code reste écrit en un Python très lisible, surtout si on est habitué à utiliser Numpy. Il en est de même du parallélisme sur processeur multicœur qui est beaucoup plus simple à écrire que si on utilise le module standard multiprocessing.
Concernant la programmation Cuda (si on dispose d’une carte graphique Nvidia), l’intérêt est qu’on a accès à la puissance de calcul d’un GPU tout en écrivant un code Python beaucoup plus simple et de beaucoup plus haut niveau que son équivalent Cuda C++.
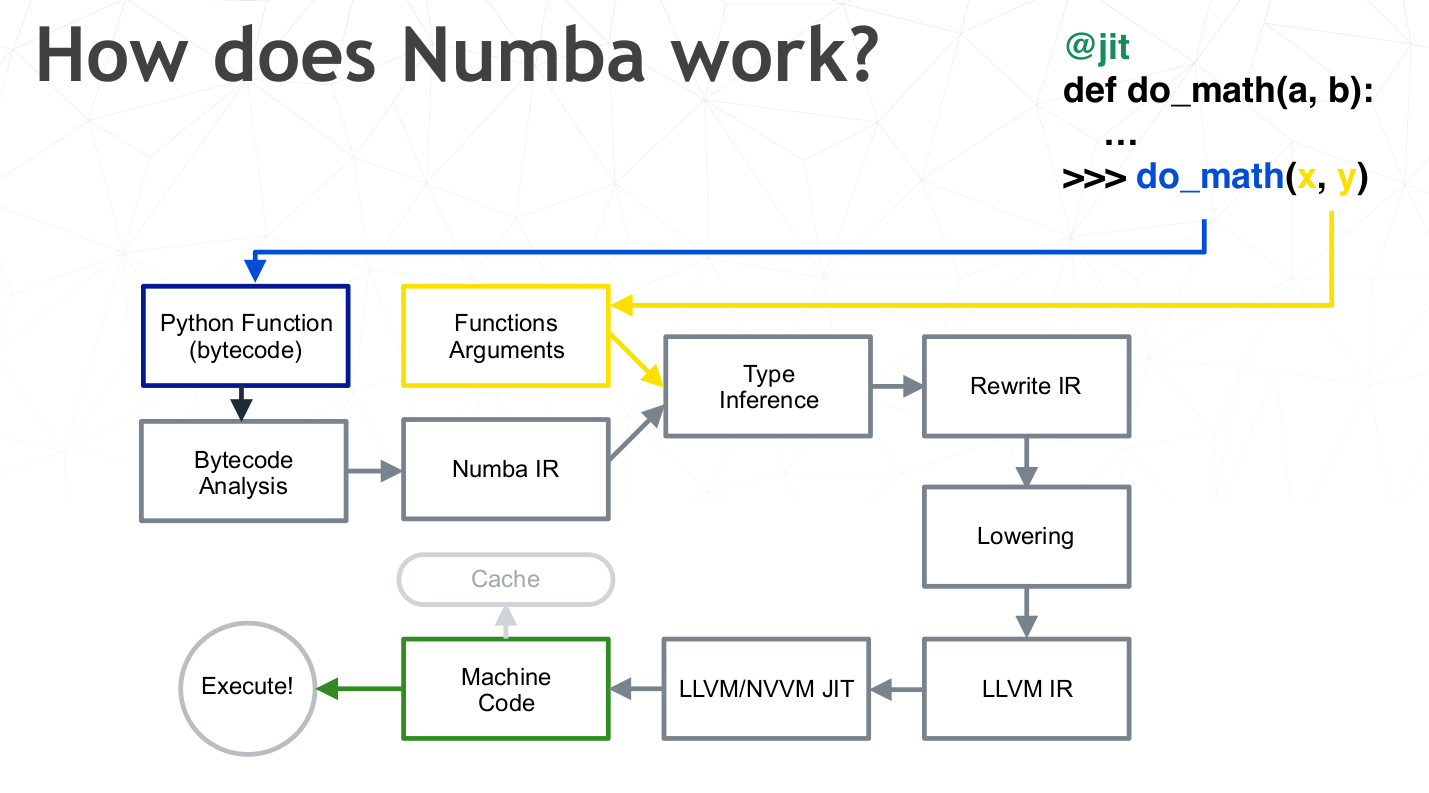
Fonctionnement interne de Numba¶
Techniquement, Numba en mode nopython est un compilateur JIT (compilation en code natif au moment de l’exécution du code). Il N’utilise PAS l”API C de Python (d’où le terme de nopython), autrement dit il ne crée pas une extension Python comme le sont ou le font la plupart des programmes qui accélèrent Python (comme Numpy, SciPy, pybind11, Cython, etc).
Numba crée en 8 étapes (et plusieurs sous étapes) son propre binaire exécutable :
- la première étape est l’analyse du bytecode Python,
- d’autres étapes servent, à partir de la connaissance ou l’inférence des types des données utilisées par la fonction à compiler, à générer une représentation intermédiaire (IR) du code, propre à Numba
- les dernières étapes consistent à envoyer cette représentation à l’infrastructure de compilateur LLVM qui en génère une représentation intermédiaire puis qui génère du code assembleur optimisé propre à la plateforme qui exécutera le code.
Ici, je me limite à utiliser Numba sous l’interpréteur CPython (le plus courant) mais il semblerait que Numba soit potentiellement utilisable sous Pypy : Running Numba on PyPy.
Limitations, inconvénients de Numba¶
L’utilisation de Numba en mode nopython entraîne de nombreuses contraintes par rapport à la programmation traditionnelle en Python. Ci-dessous, les principales que je relève
Numba n’apporte un gain de performance que sur un type limité de programmes qui utilisent des tableaux Numpy et/ou des boucles et des fonctions ou algorithmes mathématiques. Les tableaux Numpy doivent, pour être utilisables par Numba, être d’un type, d’une part, numérique (des double, des int64, etc) et, d’autre part, unique (pas de panachage de types comme une structure en C où on pourrait assembler un flottant et un int par exemple). En outre, on doit accepter la contrainte qu’un tableau Numpy ne peut contenir des tableaux Numpy de tailles différentes (ce qu’on appelle parfois des ragged array, des « tableaux en escalier »).
De très nombreuses fonctionnalités Python (et même de Numpy) ne sont pas prises en charge
Toutes les données doivent être typées. Le moindre défaut, la moindre incohérence ou ambiguïté de typage entraîne un rejet du programme avec des messages d’erreur assez cryptiques
Efficacité très inégale des structures de données de Numba autres que les tableaux Numpy, les listes Numba étant souvent lentes
La conséquence de l’inefficacité (variable) des Listes Numba est qu’on ne dispose pas de listes extensibles en mode nopython (comme les listes habituelless de Python ou les vector de la STL du C++), ce qui est très pénalisant dans de nombreuses formes d’algorithmes
Attente de la phase de compilation avant d’obtenir la bonne performance
Pas mal de bugs même si la communauté est très réactive
Évolution rapide des versions donc nécessité de souvent mettre à jour (pour suivre l’évolution des versions, consulter ICI)
Communauté d’utilisateurs assez réduite : en mai 2021, sur Stack Overflow, je compte
- 1541 questions Numba
- 4523 pour Cython,
- 18122 pour SciPy
- 89949 pour Numpy.
Me semble assez peu utilisé en production, peut-être utilisé en prototypage.
Ressources pour Numba¶
- La documentation de la dernière version au format Readthedocs
- La documentation de la dernière version au format pdf
- Description des nouveautés et améliorations de chaque version, y compris la dernière disponible.
- Présentations pdf de Numba d’avril 2018 mais riche en information et toujours d’actualité, par Stanley Seibert, ex-manager de Numba.
- Dépôt Github de Numba.
- Questions Numba sur Stack Overflow
- Portail de la mailing list Numba sur Google groups
Présentations vidéo de Numba¶
(Vérifié en mai 2021)
- Numba - Stanley Seibert, mai 2019, par le directeur de l’innovation à Anaconda (qui développe Numba)
- How to Accelerate an Existing Codebase with Numba par Stanley Seibert, SciPy 2019
- Accelerating Scientific Workloads with Numba par Siu Kwan Lam, un des principaux développeurs de Numba (avril 2018).
- Vidéo (2019) très pédagogique sur la programmation GPU : High-Performance Computing with Python: Numba and GPUs
Essayer Numba sur le site mybinder¶
Pour découvrir et essayer Numba, vous pouvez le faire en ligne sur le site mybinder. Il y a une limitation : les GPU ne sont pas disponibles (pas en libre accès en tous cas).
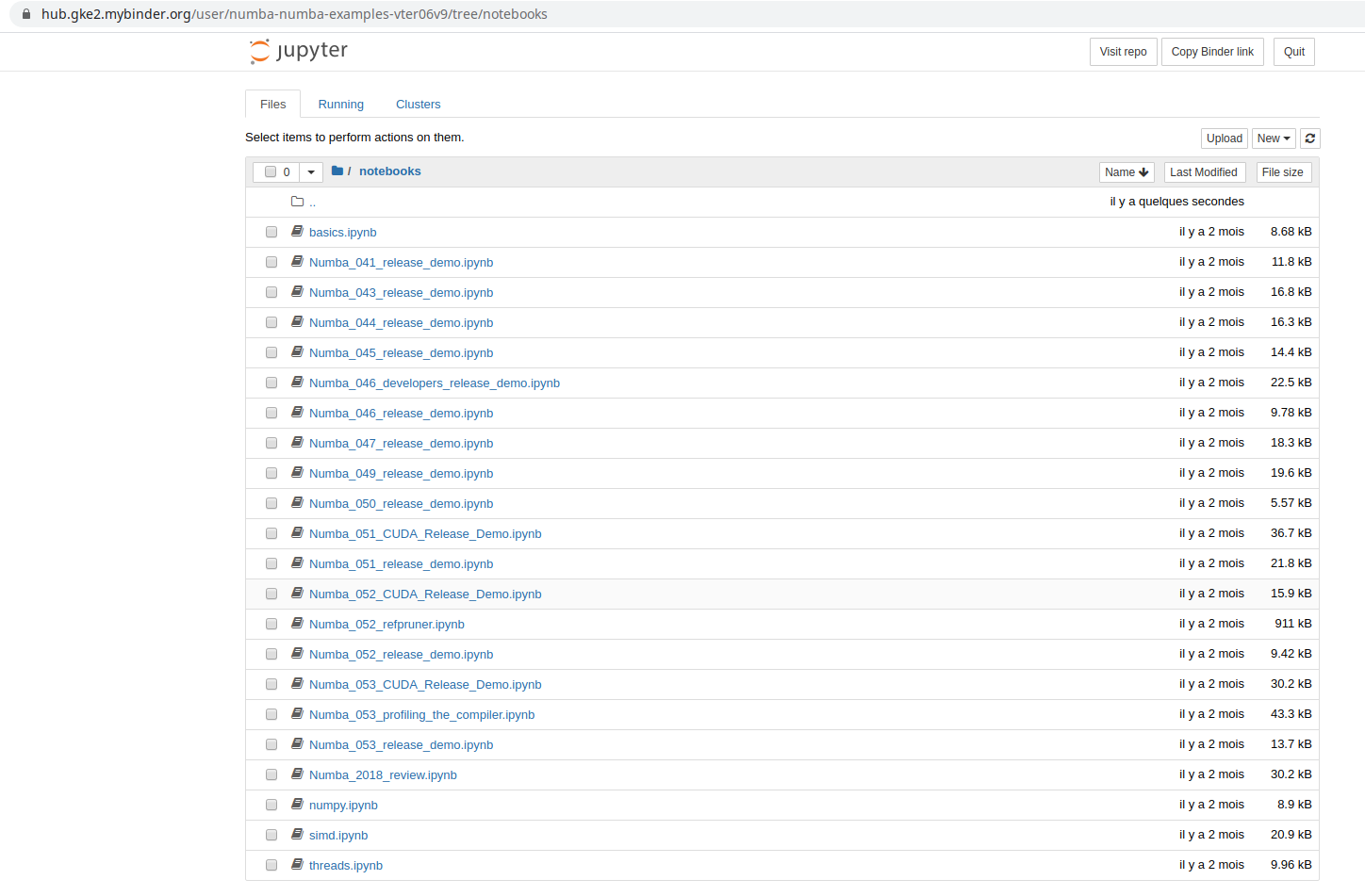
Après avoir cliqué sur le lien, attendez que l’environnement Jupyter Notebook se charge. Vous pouvez alors examiner les feuilles d’exemples proposés :
ou créer votre propre feuille Jupyter Notebook en cliquant sur New. Vous pouvez aussi uploader votre propre feuille Jupyter.
La dernière version de Numba est proposée. Les temps d’exécution sont relativement lents par rapport à ce que donne ma propre machine munie d’un processeur i5 de 2015.
L’exécutable numba¶
Quand le module Numba est installé un exécutable en ligne de commande du nom de numba est également installé. Il est destiné à des utilisateurs avancés et qui cherchent à examiner les différentes phases lors du processus de compilation (annotation du bytecode, code assembleur LLVM généré, etc).
L’exécutable numba dispose d’une option généraliste, l’option -s, qui donne des informations sur le système et qui sont utilisées par Numba. Voici un extrait (mai 2021):
$ numba -s
System info:
---------------------------------------------------------------------
...
__Hardware Information__
Machine : x86_64
CPU Name : haswell
CPU Count : 4
...
CPU Features : 64bit aes avx avx2 bmi bmi2 cmov
cx16 cx8 f16c fma fsgsbase fxsr
invpcid lzcnt mmx movbe pclmul
popcnt rdrnd sahf sse sse2 sse3
sse4.1 sse4.2 ssse3 xsave xsaveopt
...
Memory Available (MB) : 3625
__OS Information__
Platform Name : Linux-4.15.0-117-generic-x86_64-with-Ubuntu-18.04-bionic
...
Libc Version : glibc 2.25
__Python Information__
Python Compiler : GCC 8.4.0
Python Implementation : CPython
Python Version : 3.6.9
Python Locale : fr_FR.UTF-8
__LLVM Information__
LLVM Version : 10.0.1
__CUDA Information__
CUDA Device Initialized : True
CUDA Driver Version : 10020
CUDA Detect Output:
Found 1 CUDA devices
id 0 b'GeForce GTX 970' [SUPPORTED]
compute capability: 5.2
pci device id: 0
pci bus id: 1
CUDA Libraries Test Output:
Finding cublas from System
named libcublas.so
trying to open library... ok
...
__Installed Packages__
...
Informations diverses¶
- Le développement du package Numba est réalisé sous l’égide de l’entreprise Anaconda.
- Numba a été créé en 2012 par Travis Oliphant, qui est le fondateur de la bibliothèque Numpy et le créateur de Continuum Analytics, devenu par la suite Anaconda.
- Le code source de Numpy est essentiellement écrit en C et en Python. Le package llvmlite fait la liaison entre Numba et une petite partie, enveloppée en C, de l’API C++ de la bibliothèque LLVM. La liaison avec Python est assurée par le module ctypes.
- On lit de-ci de-là, que PyCuda serait plus rapide que Numba. Cela semble basé sur d’anciens benchmarks comparant NumbaPro (ancêtre de Numba) et PyCuda.
- Un article de Lena Olden en 2020 semblerait indiquer que Numba serait moins performant que d’utiliser directement Cuda en C. L’article n’est pas disponible mais l’autrice expose ses résultats dans cette vidéo : Lessons learned from comparing Numba-CUDA and C-CUDA.